
บท8 เรื่องกรดนิวคลีอิค และดีเอ็นเอ
หัวข้อของบทเรียน
นิวคลีโไซด์และนิวคลีโอไทด์
โครงสร้างดีเอ็นเอ
การจัดเรียงตัวของดีเอ็นเอและโปรตีนในโครโมโซมของสิ่งมีชีวิตพวกยูคาริโอท
รูปร่างของโครโมโซม
การลอกแบบสารพันธุกรรม
The Genetic Code and Its Translation
ดีเอ็นเอ มีชื่อเต็มว่า deoxyribonucleic acid คือ กรดดีออกซีไรโบนิวคลีอิค และ อาร์เอ็นเอ มีชื่อเต็มว่า ribonucleic acid คือ กรดไรโบนิวคลีอิค
กรดนิวคลีอิคทั้งสองชนิดประกอบด้วย มอโนนิวคลีโอไทด์ (mononucleotide) หลายๆ หน่วยมาต่อกัน มอโนนิวคลีโอไทด์จะประกอบด้วย น้ำตาลดีออกซีไรโบสในดีเอ็นเอ และ ไรโบสในอาร์เอ็นเอ ไนโตรจีนัสเบส (nitrogenous base) และกรดฟอสโฟริก
ไนโตรจีนัสเบสที่พบในธรรมชาติแบ่งเป็นสองกลุ่มคือกลุ่มแรกได้แก่ เบสพิริมิดีน (pyrimidine) มีอนุพันธ์ที่พบบ่อยอยู่สามชนิดด้วยกันคือ ไซโตซีน (cytosine) ยูราซิล (uracil) และไทมีน
(thymine) ส่วนกลุ่มที่สองได้แก่ เบสเพียวรีน (purine) ประกอบด้วย วงแหวนพิริมิดีนเชื่อมกับวงแหวนอิมิดาโซล (imidazole) มีอนุพันธ์ที่พบบ่อยอยู่สองชนิดด้วยกัน คือ อะดีนีน (adenine) และ กวานีน (guanine) สูตรโครงสร้างของเบสเพียวรีน
พิริมิดีน และอนุพันธ์ของมัน ได้แสดงไว้ใน
1.นิวคลีโอไซด์และนิวคลีโอไทด
์ [บน]
นิวคลีโอไทด์ประกอบด้วยเบสเพียวรีนและพิริมิดีนเชื่อมกับน้ำตาลเพ็นโทสด้วยพันธะเบต้า ไกลโคซิดิค น้ำตาลเพ็นโทส ได้แก่ น้ำตาลไรโบส และ
ดีออกซีไรโบส
เมื่อน้ำตาลเชื่อมกับเบสตำแหน่งคาร์บอนอะตอมของน้ำตาลจึงเป็นเลขที่มีขีดอยู่ด้วย (prime number) เพื่อให้แตกต่างจากการบอกตำแหน่งอะตอมในองค์ประกอบของเบส ตัวอย่างเช่น 2-deoxyribose ตำแหน่งคาร์บอนตัวที่สองในโมเลกุลของน้ำตาล deoxyribose คือ 2 นั่นเอง พันธะไกลโคซิดิคระหว่างน้ำตาลกับเบสเป็นพันธะที่เชื่อมต่อระหว่าง C-1 ของน้ำตาลกับ N-1 ของเบสพิริมิดีน หรือ N-9 ของเพียวรีน ตัวอย่างไรโบนิวคลีโอไซด์ได้แก่ อะดีโนซีน (adenosine) กวาโนซีน (guanosine) ไซติดีน (cytidine) ยูริดีน (uridine) และ ไทมิดีน (thymidine) ดังตารางที่1 ที่เกิดจากน้ำตาลไรโบสเชื่อมเบสอะดินีน กวานีน ไซโตซีน ยูราซิล และไทมีน ด้วยพันธะไกลโคซิดิค ตามลำดับ ตัวอย่าง ดีออกซีไรโบนิวคลีโอไซด์ ได้แก่ ดีออกซีอะดีโนซีน (deoxyadenosine) ที่เกิดจากน้ำตาลดีออกซีไรโบส เชื่อมกับเบสอะดินีนด้วยพันธะไกลโคซิดิค นิวคลีโอไซด์เกิดขึ้นจากการไฮโดรไลซ์บางส่วน (partial hydrolysis) ของนิวคลีโอไทด์ นิวคลีโอไทด์มีการสลายหมู่ฟอสเฟทออกไปจะได้นิวคลีโอไซด์ นิวคลีโอไซด์ละลายน้ำได้ดีกว่าเบสของมัน
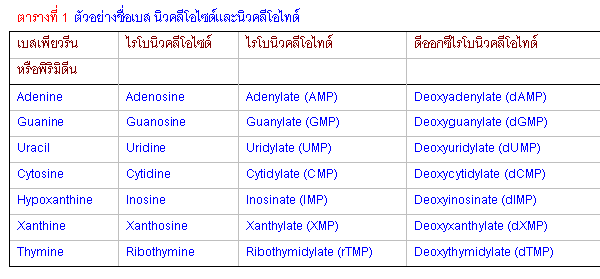 |
นิวคลีโอไทด์หน่วยเดียวจะเรียกว่ามอโนนิวคลีโอไทด์ (mononucleotide) มอโน-นิวคลีโอไทด์ 2
หน่วยมาเชื่อมกันด้วยพันธะฟอสโฟไดเอสเตอร์ระหว่างตำแหน่ง 3 ของ D-ไรโบส หรือ 2 ดีออกซี-D-ไรโบส ของมอโนนิวคลีโอไทด์หน่วยที่หนึ่งกับตำแหน่ง 5 ของมอโนนิวคลีโอไทด์หน่วยที่สอง จะเรียกว่าไดนิ
วคลีโอไทด์ (dinucleotide) ถ้ามีมอโนนิวคลีโอไทด์มากกว่าสองหน่วยมาเชื่อมต่อกันด้วยพันธะฟอสโฟไดเอสเตอร์จะเรียกว่าพอลีนิวคลีโอไทด์ (polynucleotide) พอลีนิวคลีโอไทด์ที่สำคัญคือ ดีเอ็นเอและอาร์เอ็นเอ
2.โครงสร้างของดีเอ็นเอ [บน]
ดีเอ็นเอเป็นสารที่เก็บข้อมูลทางพันธุกรรมที่พบในเซลล์โปรคาริโอทและยูคาริโอท ดีเอ็นเอขอ
งโปรคาริโอทมีลักษณะโมเลกุลเป็นวงแหวนสายคู่ไม่อยู่รวมกับโปรตีนมโมเลกุลของดีเอ็นเอในยูคาริโอทเป็นเกลียวสายคู่ (double stranded helix) อยู่รวมกับโปรตีนชนิดหนึ่งเรียกว่า ฮีสโตน (histone) จับ
กันด้วยพันธะไอออนิก ดีเอ็นเอที่อยู่รวมกับโปรตีนนี้เรียกว่า โครมาทิน (chromatin) ซึ่งอยู่ในนิวเคลียสของเซลล์ นอกจากนี้ยังสามารถพบดีเอ็นเอในไมโทคอนเดรียและใน คลอโรพลาสต์ของพวกยูคาริโอท ซึ่
งมีลักษณะเป็นวงแหวนสายคู่และไม่อยู่รวมกับโปรตีน ลักษณะเช่นนี้จะเหมือนกับดีเอ็นเอในโปรคาริโอท สำหรับไวรัสนั้นมีกรดนิวคลีอิคเป็นสารพันธุกรรมเช่นกัน อาจเป็นไปได้ทั้งดีเอ็นเอ หรืออาร์เอ็นเอ แต่มีขนาดเล็กกว่าดีเอ็นเอของแบคทีเรีย
Nature of DNA
ในปี ค.ศ. 1953 Watson และ Crick ได้เผยแพร่รายงานที่เสนอรูปแบบโครงสร้างของโมเลกุล DNA คือ DNA ประกอบด้วย 2 สายของ polynucleotide พันรอบตัวเองแบบเวียนขวาหรือตามเข็มน
าฬิกา Watson และ Crick ได้กล่าวถึง ปรากฏการณ์ที่ทำให้โครงสร้างนี้เป็นจริงไว้ 3 กรณีด้วยกันคือ
1.โมเลกุลของ DNA ประกอบด้วย base น้ำตาล และหมู่ฟอสเฟต เชื่อมต่อกันเป็นสายของ polynucleotide (รูปที่ 5)
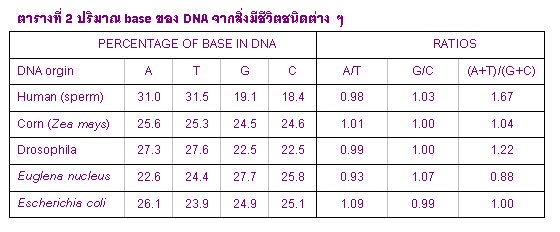
2.Erwin Chargaff ได้วิเคราะห์ DNA จากสิ่งมีชีวิตหลายชนิดพบว่าปริมาณของ purines และ pyrimidines ของ DNA ทุกชนิดจะเท่ากันดังแสดงในตารางที่ 1 นอกจากนี้ยังได้แสดงว่าอัตราส่วนของ
A+T/G+C จะเป็นค่าคงที่ในสิ่งมีชีวิตแต่ละชนิดอีกด้วย
 |
3.Rosalind Franklin และ Wilkins ได้ศึกษาจากเส้นใยของ DNA โดยใช้วิธี X-ray diffraction และบันทึกภาพของการหักเหของลำแสงของรังสี X ไว้บนฟิลม์ถ่ายภาพ (รูปที่ 6) พบว่าโมเลกุลของ DNA มีการจัดเรียงลำดับที่แน่นอนและอย่างมีระเบียบมากอยู่ในโครงสร้างที่เป็นเส้นสายที่พันกันเป็นเกลียวประกอบด้วย repeating unit ซึ่งมีช่องว่างห่างกันประมาณ 0.34 nm และ 3.4 nm ไปตามความยาวของแกนโมเลกุล
Watson และ Crick ได้รวบรวมข้อมูลทั้งหมดมาจัดทำโครงสร้าง DNA ในรูปทรง 3 มิติซึ่งพอจะสรุปได้ดังนี้
1.โมเลกุลของ DNA ประกอบด้วย 2 สายของ polynucleotide พันกันเป็นเกลียวซึ่งกันและกันในทิศวนขวาหรือตามเข็มนาฬิกา
2.มีเส้นผ่านศูนย์กลางของทรงกระบอกเป็น 2 nm
3.สายทั้ง 2 ของ polynucleotide จะขนานกันและมีทิศทางตรงกันข้ามโดยสายหนึ่งจะมีทิศจาก 5' ไป 3' ส่วนอีกสายหนึ่งจะมีทิศจาก 3' ไป 5' โดยที่ปลาย 5' มีหมู่ฟอสเฟตและที่ปลาย 3' มีหมู่ไฮดรอกซิล
4.สายของน้ำตาลที่ต่อกับหมู่ฟอสเฟตเป็น back bone นั้นจะอยู่ด้านนอกของ double helix ในขณะที่ base จัดเรียงอยู่ในส่วนกลางของแกนโดย base ของทั้ง 2 สายจัดเรียงอยู่ในโครงสร้างแบบราบและตั้ง
ฉากกับแกนของโมเลกุล เปรียบเสมือนขั้นบันไดที่อยู่ระหว่างราวบันไดที่เวียนขวา
5.base ของทั้ง 2 สายของ polynucleotide จะยึดกันด้วยพันธะไฮโดรเจนและมีการจับคู่กันอย่างเฉพ
าะเจาะจงคือ A=T โดย 2 พันธะไฮโดรเจนและ G=C โดย 3 พันธะไฮโดรเจน เรียกว่า complementary base pair (รูปที่ 7) ทำให้โมเลกุลของ DNA ง่ายต่อการที่จะแยกจากกันเป็น 2 สาย
ดังนั้นในการสังเคราะห์ DNA โมเลกุลใหม่โดยกลไกลที่เรียกว่า semiconservative replication ทำให้ลำดับของนิวคลีโอไทด์บนสายเก่าจะเป็นตัวกำหนดลำดับของนิวคลีโอไทด์บนสายใหม่ โดยอาศัยกฎของ
ของ complementary base pair ทำให้ได้ลำดับของนิวคลีโอไทด์ทั้ง 2 สายบน DNA โมเลกุลใหม่ ทั้ง 2 โมเลกุลเหมือนกันและเหมือนโมเลกุลเดิม
6.คู่ base จะอยู่ห่างประมาณ 0.34 nm ดังนั้น 1 รอบของการหมุน 360o มีความยาว 3.4 nm มี 10 คู่
base ดังนั้นแต่ละคู่จะเอียงเป็นมุม 36o
7.เนื่องจากระยะระหว่างพันธะของ phosphodiester ของ back bone ในแต่ละสายมีตำแหน่งไม่ตรง
กัน จึงเป็นผลทำให้เกิดเป็นช่องว่างที่ไม่เท่ากันเรียกว่า major groove (wide groove) และ minor groove (narrower groove) ดังแสดงในรูปที่ 8
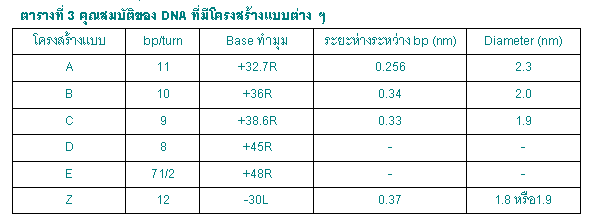
นอกจากโครงสร้าง DNA แบบที่เสนอโดย Watson และ Crick ซึ่งเรียกว่าแบบ B แล้วต่อมายังมีการค้นพบโครงสร้างแบบต่าง ๆ ของ DNA อีกดังแสดงในตารางที่ 3
 |
ดีเอ็นเอของยูคาริโอทส่วนใหญ่ (98%) จะพบในนิวเคลียส มีโครงสร้างเป็นสายพอลีนิวคลีโอไท
ด์สองสายที่กลับทิศทางกัน (antiparallel) พันกันเป็นเกลียวคู่ (double helix) โดยมีเบสเพียวรีนและพิริมิดีนอยู่ใกล้กันและตั้งฉากกับน้ำตาล-ฟอสเฟท โครงสร้างกระดูกสันหลังของดีเอ็นเอนั้นสร้างพันธะไฮโด
รเจนเชื่อมระหว่างเบสคู่สม (complementary bases) ทำให้พอลินิวคลีโอไทด์สองสายนั้นจับกันอย่างจำเพาะด้วยพันธะไฮโดรเจน การจับคู่สมของเบส (complementary base pairing) อย่างจำเพาะระหว่
างอะดินีนจับคู่กับไทมีนและกวานีนจับคู่กับไซโตซีน ความรู้นี้ได้จากการวิเคราะห์โครงสร้างของดีเอ็นเอด้วยวิธีการหักเหกระจายของรังสีเอ็กซ์ (x-ray diffraction)โดยนักพันธุศาสตร์ชาวอเมริกาชื่อ เจมส์ วัตสั
น (James Watson) และนักฟิสิกส์ชาวอังกฤษชื่อ ฟรานซิส คริก (Francis Crick) ในปี ค.ศ. 1953 วัตสันและคริกได้เสนอโครงสร้างดีเอ็นเอเป็นเกลียวคู่ ประกอบด้วยสายพอลินิวคลีโอไทด์ 2 สายมีทิศทางพัน
กันในลักษณะเป็นเกลียววนขวา เรียกว่า ดีเอ็นเอแบบบี (มีเส้นผ่านศูนย์กลาประมาณ 20 อังสตรอม (Ao) มีระยะหนึ่งรอบเกลียวเท่ากับ 3.4 nm หรือ 34 Ao มีเบสคู่สมราวสิบคู่ต่อรอบ เบสเพียวรีนและพิริมิดีน
อยู่ในเกลียวในลักษณะที่ซ้อนขนานกัน มีระนาบเกือบตั้งได้ฉากกับแกนของเกลียว เกลียวนี้มีร่องใหญ่ (major groove) และร่องเล็ก (minor groove) ตลอดเกลียว นอกจากดีเอ็นเอแบบบีแล้ว ยังพบดีเอ็นเ
อแบบเอ และ ซี ซึ่งต่างก็เป็นเกลียวคู่เวียนขวาคล้ายๆกัน ปัจจุบันยังได้พบดีเอ็นเอแบบแซด (Z-DNA) ซึ่งเป็นเกลียวคู่เวียนซ้าย ดีเอ็นเอจะมีโครงสร้างแบบใดมักขึ้นอยู่กับชนิดของเบส ปริมาณน้ำและไอออนโลหะที่จับคู่อยู่กับดีเอ็นเอ
3 การจัดเรียงตัวของดีเอ็นเอและโปรตีนในโครโมโซมของสิ่งมีชีวิตพวกยูคาริโอท
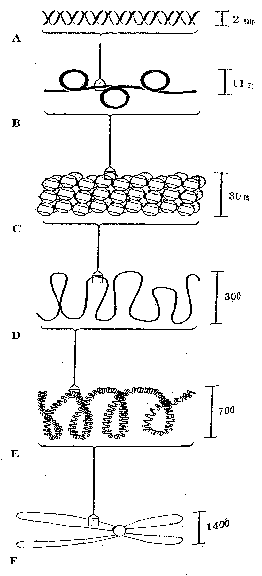
สายโครมาตินขณะที่ยังไม่มีการพันเกลียวเกิดขึ้นจะมีเส้นผ่าศูนย์กลาง 11 นาโนเมตร เมื่อเกิดการพันเกลียวและสั้นขึ้นจะทำให้ได้สายโครมาตินที่มีเส้นผ่าศูนย์กลางใหญ่ขึ้นเป็น 300 อังสตรอม หรือ 30
นาโนเมตร หรือที่เรียกว่า โซลีนอยด์ (solenoid) ซึ่งการพันเกลียวแต่ละรอบจะมีนิวคลีโอโซมประมาณ 6 นิวคลีโอโซม หลังจากนั้นสายโครมาตินที่มีเส้นผ่าศูนย์กลาง 30 นาโนเมตร หรือโซลีนอยด์นี้จะเกิดการ
พันเกลียวต่อไป และมีการม้วนตัวจนเกิดห่วงขึ้นเป็นจำนวนมาก และทำให้มีเส้นผ่าศูนย์กลางใหญ่ขึ้นจากเดิมเป็น 300 นาโนเมตร และจะมีการพันเกลียวต่อไปจนได้เส้นผ่าศูนย์กลางใหญ่ขึ้นเป็น 700 นาโนเ
มตร ซึ่งจะกลายเป็นโครมาติดของโครโมโซมในระยะเมทาเฟสนั่นเอง ดังนั้น โครโมโซมในระยะเมท
าเฟสจะมีเส้นผ่าศูนย์กลาง 1400 นาโนเมตร [บน]
ดูรูปที่ 4
เซลล์พวกยูคาริโอทนอกจากจะมีดีเอ็นเอในนิวเคลียสแล้ว ยังมีดีเอ็นเอปริมาณน้อยอยู่ในไมโทคอนเ
ดรียและในคลอโรพลาสต์ของเซลล์ที่สังเคราะห์แสงคิดเป็นประมาณ 0.1% ของดีเอ็นเอทั้งหมด ดีเอ็นเอในไมโทคอนเดรียและคลอโรพลาสต์จะมีขนาดเล็กเป็นวงแหวนเกลียวคู่ มีมวลโมเลกุล ประมาณ
10,000 กิโลดาลตัน และไม่อยู่รวมกับฮิสโตน ดีเอ็นเอเหล่านี้บรรจุข้อความทางพันธุกรรมที่จะถูกถ่ายทอดมาเป็นอาร์เอ็นเอ และโปรตีนในไมโทคอนเดรีย และคลอโรพลาสต์ อย่างไรก็ตามโปรตีนอื่นๆในไมโ
ทคอนเดรีย ประมาณ 95% จะถูกสร้างมาจากดีเอ็นเอในนิวเคลียส ตัวอย่างการศึกษาไมโทคอนเดรียของยีสต์ พบว่า ไซโตโครมในเมมเบรนของไมโทคอนเดรียสร้างมาจากยีนในดีเอ็นเอของไมโทคอนเดรียร่
วมกับยืนในดีเอ็นเอของนิวเคลียส โปรตีนนี้มีความสำคัญในห่วงโซ่หายใจ (respiratory chain) ของเซลล์
 |
4 รูปร่างของโครโมโซม
[บน]
สิ่งมีชีวิตแต่ละชนิดจะมีจำนวนโครโมโซมที่แน่นอน และโครโมโซมแต่ละแท่งจะมีขนาดและรูปร่า
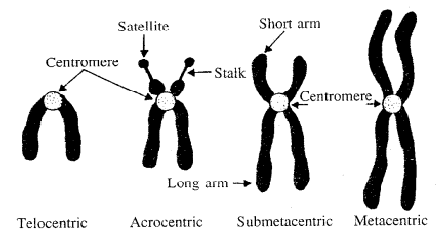
งคงที่ด้วย โดยในสิ่งมีชีวิตชนิดหนึ่งอาจมีโครโมโซมที่มีรูปร่างแบบเดียวหรือหลายแบบก็ได้ถ้าใช้ตำแหน่งเซนโทรเมียร์บนโครโมโซมเป็นหลัก สามารถจะแบ่งโครโมโซมออกได้เป็น 4 แบบคือ
1.เมทาเซนทริก โครโมโซม (metacentric chromosome) คือ โครโมโซมที่มีตำแหน่งเซนโทรเมียร์อยู่ตรงกลาง ทำให้แขนทั้ง 2 ข้างของโครโมโซมยาวเท่ากัน เมื่อโครโมโซมชนิดนี้ถูกดึงเข้าสู่ขั้วเซลล์ร
ะหว่างที่มีการแบ่งเซลล์ จะเห็นโครโมโซมเป็นรูปตัววี (v shape)
2.ซับเมทาเซนทริก โครโมโซม (submetacentric chromosome) คือ โครโมโซมที่มีตำแหน่งเซนโ
ทรเมียร์อยู่ใกล้กับตำแหน่งตรงกลางโครโมโซม ทำให้มีแขนข้างหนึ่งสั้น และแขนอีกข้างหนึ่งยาว เมื่อโครโมโซมชนิดนี้ถูกดึงเข้าสู่ขั้วเซลล์ระหว่างที่มีการแบ่งเซลล์ จะเห็นโครโมโซมเป็นรูปตัวเจ (j shape)
3.เอโครเซนทริกโครโมโซม (acrocentric chromosome) คือ โครโมโซมที่มีตำแหน่งเซนโทรเมียร์อยู่ค่อนไปทางปลายใดปลายหนึ่งของโครโมโซม ทำให้มีแขนข้างหนึ่งสั้นและแขนอีกข้างหนึ่งยาวเช่นเดีย
วกัน เมื่อโครโมโซมชนิดนี้ถูกถึงเข้าสู่ขั้วเซลล์ จะเห็นโครโมโซมเป็นรูปตัวเจ (j shape)
4.เทโลเซนทริกโครโมโซม (telocentric chromosome) คือ โครโมโซมที่มีตำแหน่ง
เซนโทรเมียร์อยู่ที่ปลายข้างหนึ่งของโครโมโซม ทำให้มีแขนเพียงข้างเดียว เมื่อโครโมโซมชนิดนี้ถูกดึงเข้าสู่ขั้วเซลล์ จะเห็นโครโมโซมเป็นรูปแท่ง (rod shape) (รูปที 5)
ปกติรูปร่างของโครโมโซมเหล่านี้เปลี่ยนแปลงน้อยมาก แต่บางครั้งถ้าโครโมโซมเหล่านี้กระทบกับรังสีต่างๆ เช่น รังสีเอกซ์ รังสีคอสมิก รังสีแกมมา หรือสารเคมีต่างๆ อาจทำให้โครโมโซมหรือโครมาติดเกิด
การแตกหักได้ ทำให้เกิดปลายเหนียว (sticky end) ขึ้น 2 ด้าน ซึ่งมีผลทำให้โครงสร้างของโครโมโซมมีโอกาสเปลี่ยนแปลงไปได้ 3 ทาง คือ
1.เกิดการขาดหายไปของชิ้นส่วนของโครโมโซมที่ไม่มีเซนโทรเมียร์
 |
2.เกิดการต่อกันใหม่ของชิ้นส่วนของโครโมโซมที่ขาดออกจากกัน ทำให้ได้โครงสร้างของโครโมโซมเหมือนเดิม
3.ปลายที่แตกหักปลายหนึ่งหรือทั้งสองปลาย อาจจะไปต่อกับปลายที่แตกหัก ตำแหน่งอื่นบนโครโมโซมเดียวกัน หรือต่อกับโครโมโซมต่างคู่กันได้ ทำให้เกิดการแลกเปลี่ยนชิ้นส่วนของโครโมโซมต่างคู่กัน
5.การลอกแบบสารพันธุกรรม
[บน]
การลอกแบบสารพันธุกรรมหรือข้อความทางพันธุกรรม (replication) คือ การสังเคราะห์ดีเอ็นเอให
ม่ให้เหมือนดีเอ็นเอคู่เดิมเป็นกระบวนการลอกแบบสารพันธุกรรมให้ดีเอ็นเอที่สังเคราะห์ขึ้นมาใหม่มีลำดับการเรียงตัวของนิวคลีโอไทด์เหมือนดีเอ็นเอเดิมที่ใช้เป็นแม่แบบ (template) เมื่อพิจารณาถึงโอกา
สที่จะเป็นไปได้ในกรณีนี้มี 3 วิธีดังนี้คือ
* วิธีอนุรักษ์ (conservative) คือการที่ดีเอ็นเอเดิมเป็นแม่พิมพ์โดยสร้างดีเอ็นเอใหม่หนึ่งคู่ให้เหมือนดีเอ็นเอเดิม
* วิธีกึ่งอนุรักษ์ (semiconservative) คือการที่ดีเอ็นเอคู่เดิมถูกแยกออกเป็นสายเดี่ยว (single strand) แล้วเกิดการสังเคราะห์ดีเอ็นเอสายใหม่มาประกบเข้าคู่กับสายเก่า โดยอาศัยกฎการจับคู่ของเบ
ส ผลที่ได้คือ ดีเอ็นเอสองคู่แต่ละคู่ประกอบด้วยดีเอ็นเอเดิมหนึ่งสาย และดีเอ็นเอใหม่อีกหนึ่งสาย จึงเป็นการคงดีเอ็นเอเดิมไว้เพียงครึ่งเดียว
* วิธีกระจาย (dipersive) คือการที่ดีเอ็นเอคู่เดิมถูกตัดออกเป็นช่วงๆ แล้วแยกออกเป็นสองส่วน แต่ละส่วนก็จะมีดีเอ็นเอใหม่สร้างขึ้นมาซ่อมแซมส่วนที่ขาดหายไปจนครบสมบูรณ์ ผลที่ได้คือ ดีเอ็นเอใหม่ทั้งสอ
งคู่จะมีส่วนของดีเอ็นเอเก่าอยู่กระจัดกระจายตลอดเกลียวคู่
ในกระบวนการจำลองโมเลกุลของดีเอ็นเอเป็นแบบวิธีกึ่งอนุรักษ์ โดยเริ่มต้นจากโมเลกุลเดิมของดีเ
อ็นเอที่ประกอบด้วยโพลีนิวคลีโอไทด์ 2 สายทิศทางสวนกันและขนานกัน โพลีนิวคลีโอไทด์แต่ละสายจะทำหน้าที่เป็นแม่แบบเพื่อจะสร้างโพลีนิวคลีโอไทด์สายใหม่ขึ้นมาเข้าคู่ ที่เรียกว่า เป็นแบบกึ่งอนุรักษ์นั้น อ
ย่างไรก็ตามทิศทางของการสร้างโพลีนิวคลีโอไทด์สายใหม่นั้นจะมีทิศทางเป็นแบบ 5 -> 3 เสมอ ดังนั้นในการจำลองโมเลกุลของดีเอ็นเอจะมีการสร้างโพลีนิวคลีโอไทด์สายใหม่สายหนึ่งแบบยาวตลอดเรียกส
ายนี้ว่า สายลีดดิง (leading strand) สำหรับโพลีนิวคลีโอไทด์สายใหม่อีกสายหนึ่งไม่สามารถสร้างต่อเนื่องกันเป็นสายยาวตลอดไปได้ เพราะทิศทางการสร้างสวนกับทิศทางการคลายเกลียวของโมเลกุลเดิมขอ
งดีเอ็นเอ จึงมีการสร้างโพลีนิวคลีโอไทด์สายสั้นๆ ขึ้นมาก่อนแล้วจึงมาต่อกันเป็นสายยาว เพื่อให้ทิศทางการสร้างเป็นแบบ 5 -> 3 เช่นกัน เรียกสายนี้ว่า สายแลกกิง (lagging strand) โพลีนิวคลีโอไทด์สายสั้
นๆนี้ ประกอบด้วยนิวคลีโอไทด์ประมาณ 1,000 ถึง 2,000 นิวคลีโอไทด์ เรียกโพลีนิวคลีโอไทด์สายสั้นๆนี้ว่า โอคาซากิ แฟรกเมนท์ (Okazaki fragments) ซึ่งเป็นการให้เกียรติกับผู้ค้นพบคือ ไรจิ โอคาซากิ (Reiji Okazaki)
สำหรับรายละเอียดกระบวนการจำลองโมเลกุลของดีเอ็นเอ เริ่มต้นด้วยมีเอนไซม์เฮลิเคส ทำหน้าที่สลายพันธะไฮโดรเจน (H-bond) เพื่อให้เกลียวคู่ของดีเอ็นเอเริ่มแยกออกจากกัน ทำให้เกิดส่วนที่โป่งออ
กมา (bubble) จากนั้นจะมีเฮลิกซ์ ดีสเตบิไลซิงโปรตีน เข้าไปจับกับดีเอ็นเอสายเดี่ยวเพื่อป้องกันไม่ให้กลับคืนเป็นเกลียวคู่อีก และจะมีเอนไซม์ดีเอ็นเอไจเลส ทำหน้าที่คลายปมเหนือจุดแยก (replication
fork) โดยการตัดสายหนึ่งของดีเอ็นเอออก เพื่อทำให้เกิดการคลายเกลียวและทำให้เกิดการคลายปมแล้ว จึงทำหน้าที่ต่อจุดที่ตัดนั้นกลับเข้าไปดังเดิม บริเวณที่โป่งออกของสายดีเอ็นเอนั้นจะเป็นจุดเริ่มของการจ
ำลองโมเลกุล (initiation point) โดยมีเอนไซม์ไพรเมสสร้างสายอาร์เอ็นเอเริ่มต้น (RNA primer) จากนั้นจะเป็นหน้าที่ของดีเอ็นเอโพลีเมอเรส 3 ทำหน้าที่เติมนิวคลีโอไทด์ที่ปลายด้าน 3-OH ของสายอา
ร์เอ็นเอเริ่มต้นนั้นในทิศทาง 5 -> 3 ไปเรื่อยๆ ตามทิศทางของการคลายเกลียว โดยใช้ดีเอ็นเอสายเดิมเป็นแม่แบบ ก็จะได้ดีเอ็นเอสายใหม่ยาวตลอด เป็นสายลีดดิง (leading strand) สำหรับการสังเคราะ
ห์โพลีนิวคลีโอไทด์สายใหม่อีกสายหนึ่งนั้น เอนไซม์ไพรเมสจะสร้างสายอาร์เอ็นเอเริ่มต้น (RNA primer) ขึ้นมาเช่นเดียวกัน และเอนไซม์ดีเอ็นเอโพลีเมอเรส 3 จะทำหน้าที่เติมนิวคลีโอไทด์เข้าที่ปลาย
3-OH ของสายอาร์เอ็นเอเริ่มต้นนี้แต่จะได้โพลีนิวคลีโอไทด์ของดีเอ็นเอสายนั้นๆ คือ โอคาซากิ แฟรกเมนท์ (Okazaki fragment) โดยทิศทางการสังเคราะห์จะสวนกับทิศทางของการคลายเกลียวของโมเล
กุลของดีเอ็นเอเดิม แต่ทิศทางยังคงเป็นแบบ 5 -> 3 เช่นเดิม เมื่อมีการคลายเกลียวของโมเลกุลของดีเอ็นเอเดิมต่อไปอีก เอนไซม์ไพรเมสก็จะสร้างสายอาร์เอ็นเอเริ่มต้นอีก และเอนไซม์ดีเอ็นเอโพล
ีเมอเรส 3 ก็จะทำหน้าที่เติมนิวคลีโอไทด์ที่ปลาย 3-OH ของสายอาร์เอ็นเอเริ่มต้นต่อไปอีกก็จะได้สายโอคาซากิแฟรกเมนท์อีก การสังเคราะห์ดีเอ็นเอจากสายแม่แบบนี้เป็นเช่นนี้เรื่อยๆไป จึงเรียกสายนี้ว่า สา
ยแลกกิง (lagging strand) หลังจากนั้นจะเป็นหน้าที่ของเอนไซม์ดีเอ็นเอโพลีเมอเรส 1 ทำการตัดสายอาร์เอ็นเอเริ่มต้น (RNA primer) ออกไปทั้งหมด และเติมนิวคลีโอไทด์ที่ปลาย 3-OH ของสายดีเอ็นเอ
สั้นๆ หรือโอคาซากิ-แฟรกเมนท์ จนกระทั่งเหลือช่องว่างเฉพาะปลาย 3-OH และ 5-PO4 หรือที่เรียกว่า นิค (nick) เท่านั้น สุดท้ายจะเป็นหน้าที่ของเอนไซม์ดีเอ็นเอไลเกส (DNA ligase) ที่จะเชื่อมปลาย 3-OH และ 5-PO4 เข้าด้วยกันด้วยพันธะเอสเตอร์ (ester bond)ก็จะได้สายดีเอ็นเอสายยาวตลอดสาย
จากการวิเคราะห์การจำลองตัวเองของ DNA โดย Kornberg และคณะ พบว่า ดีเอ็นเอพอลีเมอเรสนั้นต้องอาศัยไพรเมอร์ (primer) ซึ่งเป็นนิวคลีโอไทด์สั้นๆ สำหรับที่จะให้ดีออกซิไรโบ-นิวคลีโอไทด์ต่อเติ
มเข้าไปทางด้าน 3 ไฮดรอกซิลและอาศัยดีเอ็นเอสายหนึ่งสายใดในเกลียวคู่ เป็นดีเอ็นเอแม่แบบ โดยที่สายดีเอ็นเอที่สร้างขึ้นใหม่จะมีการเรียงตัวของเบสที่จะให้เบสทุกตัวสามารถจับคู่กับเบสในอีกสายหนึ่งข
องดีเอ็นเอแม่แบบได้ ทำให้สายดีเอ็นเอที่กำลังสร้างขึ้นใหม่มีดีออกซีไรโบ-นิวคลีโอไทด์เติมต่อไปในทิศ 3-ไฮดรอกซิล นั่นก็คือการสร้างดีเอ็นเอจะเป็นไปได้เพียงทิศเดียวคือจากปลาย 5 ไปสู่ด้าน 3 เสมอ
ปัจจุบันนี้เราทราบว่าการสังเคราะห์ดีเอ็นเอนี้ไม่ได้อาศัยดีเอ็นเอ พอลีเมอเรสเพียงอย่างเดียว แต่เป็นกระบวนการที่ซับซ้อน ต้องอาศัยเอนไซม์มากกว่า 20 ชนิด และแฟคเตอร์อื่นๆ ร่วมอยู่ด้วย โดยทำหน้าที่ปร
ะสานกันอย่างเฉพาะเจาะจงและเป็นระเบียบพอ
สรุปขั้นตอนต่างๆได้ดังนี้คือ
ก.การแยกสายดีเอ็นเอเกลียวคู่ที่เป็นแม่พิมพ์ออกจากกัน
ข.เริ่มสร้างสายดีเอ็นเอเส้นใหม่จากแม่พิมพ์ทั้งสองสายของดีเอ็นเอเดิม
ค.การเติมดีออกซีนิวคลีโอไทด์ตัวใหม่ลงไปในสายดีเอ็นเอที่กำลังสร้างอยู่
ง.การเกิดเกลียวคู่ของดีเอ็นเอใหม่
ซ.การหยุดการสังเคราะห์
The Genetic Code and Its Translation [บน]
Genes and Proteins
ในปัจจุบันนี้ตามที่ทราบกันมาแล้วว่ายีนเป็นสารประกอบทางเคมีที่อยู่ในส่วนของโครโมโซม ซึ่งบรรจุข้อมูลที่เป็นผลิตผลที่เฉพาะเจาะจงไม่ว่าจะเป็นเรื่องของโครงสร้างหรือ หน้าที่การทำงานของเซลล์ ดัง
นั้นสารพันธุกรรมที่บรรจุอยู่ใน genome ของสิ่งมีชีวิตนั้น ๆ จึงถูกเรียกว่าเป็น "blueprint for life" เนื่องจากบรรจุข้อมูลที่เกี่ยวข้องกับการทำงานของยีน ทำให้สิ่งมีชีวิตนั้น ๆ มีกิจกรรมไปด้วย การทำงานของ
ยีนเป็นอย่างไร มีความสัมพันธ์กับโปรตีนอย่างไร เกี่ยวข้องกับการแสดงออกได้อย่างไร จึงเป็นที่สนใจของนักวิทยาศาสตร์ตลอดมา ปรากฏการณ์ ต่อไปนี้เป็นข้อมูลที่ให้ทราบว่ายีนมีความสัมพันธ์กับโปรตีนอย่างไร
Inborn Errors of Metabolism
เป็นครั้งแรกที่ได้มีการศึกษาถึงหน้าที่ของโปรตีนในกระบวนการทางพันธุกรรม โดย Sir Garrod แ
ละ Bateson โดย Garrod ได้ให้ความสนใจในความผิดปกติของคน ซึ่งดูเหมือนว่าได้รับการถ่ายทอดทางพันธุกรรม ในปี ค.ศ. 1920 Garrod ได้พบปรากฎการณ์ที่แสดงถึงความสัมพันธ์ของเอนไซม์กับอากา
รผิดปกติเป็นครั้งแรก เขาได้ศึกษาความผิดปกติของคนที่เป็น alkaptonuria อาการที่แสดงออก คือ ปัสสาวะทิ้งไว้ในอากาศจะเปลี่ยนเป็นสีดำ นอกจากนี้เขายังพบว่า ครอบครัวที่เป็นโรคนี้จะถูกถ่ายทอดไป ดั
งนั้น Garrod และ Bateson จึงสรุปว่า alkaptonuria เป็นโรคพันธุกรรม เนื่องจาก
1.สมาชิกของครอบครัวหลายคนเป็น alkaptonuria
2.โรคนี้จะพบในลูกที่เกิดจากการแต่งงานระหว่างพี่น้องกัน มากกว่าการแต่งงานที่คู่แต่งงานไม่ได้เป็นญาติกัน
Garrod พบว่าคนป่วยที่เป็นโรคนี้ จะมี homogentisic acid (HA) ถูกขับออกมาทางปัสสาวะ ลักษณะ alkaptonuria เป็นลักษณะ recessive ดังนั้นคนที่ป่วยเป็นโรคนี้ จะเป็น homozygous
genotype เป็นยีนที่อยู่บน autosome คนป่วยเป็นโรคนี้ จะไม่มีเอนไซม์ที่ไปกระตุ้นให้เกิดการเปลี่ยน HA ให้เป็น Meleylacetoacetic acid จึงทำให้เกิดการคั่งของ HA Garrod เรียกความผิดปกติแบบนี้ว่า Inborn Errors of Metabolism
นอกจากนี้ Garrod ได้ศึกษาคนที่มีความผิดปกติในโรคอื่น ๆ อีก 3 โรค ทำให้เขาสามารถสรุปได้ว่า สาเหตุที่คนเหล่านี้มีความผิดปกติ เนื่องจาก metabolic pathway ถูก block ซึ่งสามารถตรวจหาส
ารเคมีที่คั่งอยู่ได้ ทำให้ทราบขั้นตอนของการถูก block ได้
อีก 40 ปีต่อมา จึงได้มีการค้นพบซึ่งเป็นการพิสูจน์ว่า ยีน และ เอนไซม์ มีความสัมพันธ์กันโดย Beadle และ Tatum
One gene-one enzyme hypothesis
ในปี ค.ศ. 1942 Beadle และ Tatum ได้ศึกษาในราพวก Neurospora crassa เพื่อใช้อธิบายถึ
ง nature of metabolic pathway โดยการทำรา Neurospora ให้เกิดมิวเตชั่น จาก prototroph (สามารถสังเคราะห์สารประกอบได้ด้วยตัวเอง) มาเป็นพวก auxotroph (ไม่สามารถสังเคราะห์สารประกอ
บได้) การเกิด auxotroph มีสาเหตุเนื่องจากกระบวนการสังเคราะห์สารประกอบต่าง ๆ มีความผิดปกติจึงทำให้ไม่สามารถสังเคราะห์สารต่าง ๆ ที่เป็นผลิตผลสุดท้ายได้หรือเนื่องจากกระบวนการสังเคราะห์สา
รเหล่านี้ถูก block เช่น กระบวนการสังเคราะห์สารประกอบ D ประกอบด้วยขั้นตอนต่าง ๆ 4 ขั้นตอนดังนี้
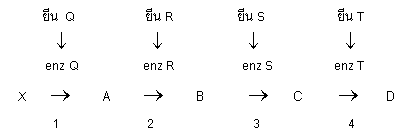 |
โปรตีนบางชนิดช่วยจับดีเอ็นเอที่แยกจากกันแล้วไม่ให้คืนกลับสู่สภาพเดิม โปรตีนนี้เรียกชื่อว่า
single-strand binding protein (SSB) นอกจากนี้ยังมีเอนไซม์ชื่อ topoisomerase ซึ่งทำหน้าที่คลายเกลียว supercoil ของดีเอ็นเอที่เกิดขึ้นหลังจากมีการแยกสายคู่ของดีเอ็นเอ การทำงานของเอนไซม์ทั้งสองนี้อาศัยพลังงานจาก ATP
นอกจากนั้น นักวิทยาศาสตร์ชาวญี่ปุ่นชื่อ Okazaki พบว่า การลอกแบบของดีเอ็นเอมีลักษณะเป็นชิ้นส่วนเล็กๆของดีเอ็นเอเกิดขึ้นหลายท่อนก่อน แต่ละชิ้นส่วนของดีเอ็นเอนี้ต่อมาเรียกว่าชิ้นส่วนโอคาซากิ
(Okazaki fragments) มีความยาวของเบสประมาณ 1,000-2,000 นิวคลีโอไทด์ในโปรคาริโอท และประมาณ 100-200 นิวคลีโอไทด์ในพวกยูคาริโอท
ในกลุ่มพวกยูคาริโอท พบว่า ความยาวของชิ้นส่วนโอคาซากิ มีเพียง 200 นิวคลีโอไทด์ ซึ่งสั้นกว่าในพวกโปรคาริโอท การศึกษาต่อมาพบว่าการสร้างชิ้นส่วนโอคาซากินั้นต้องอาศัย
อาร์เอ็นเอเป็นไพรเมอร์ (รูปที่ 3.5) ซึ่งก็คือพอลีไรโบนิวคลีโอไทด์ที่ถูกสร้างขึ้นโดยอาร์เอ็นเอ-
พอลีเมอเรสมีความยาวไม่มากนักประมาณ 1-60 นิวคลีโอไทด์ขึ้นอยู่กับชนิดของเซลล์ การสังเคราะห์ไพ
รเมอร์นี้อาศัยดีเอ็นเอสายหนึ่งเป็นแม่แบบในทิศทางการสร้างจากปลาย 5 ไปยัง 3 และต้องการ ATP, CTP, UTP และ GTP เป็นสารตั้งต้น เอนไซม์ที่ทำหน้าที่ในปฏิกิริยาการสังเคราะห์อาร์เอ็นเอไพรเมอร์
ชื่อ ไพรเมส (primase) หรือไพรเมอเรส (primerase) สำหรับการเติมดีออกซีไรโบนิวคลีโอไทด์ตัวถัดมาจากอาร์เอ็นเอไพรเมอร์นั้น อาศัยดีเอ็นเอพอลีเมอเรส ดีเอ็นเอสายใหม่ทาง lagging strand สิ้นสุด
ลง อาร์เอ็นเอไพรเมอร์จะถูกตัดออกจากชิ้นส่วนโอคาซากิทีละหนึ่งนิวคลีโอไทด์ โดยอาศัยคุณสมบัติ 5 -> 3 เอกโซนิวเคลียส (exonuclease activity) ของเอนไซม์ดีเอ็นเอพอลีเมอเรสชนิด I หรือ Rnase
H และขณะเดียวกันเอนไซม์ดีเอ็นเอพอลีเมอเรสชนิด I จะช่วยต่อเติมส่วนของนิวคลีโอไทด์ที่หายไปให้ครบเหมือนเดิม แต่ละชิ้นส่วนสั้นๆ ของดีเอ็นเอใน lagging strand จะเชื่อมต่อกัน ไฮดรอกซี (OH-) ที่ป
ลายข้างหนึ่งของดีเอ็นเอและหมู่ 5 ฟอสเฟท (PO4) ของอีกปลายหนึ่งในชิ้นส่วนโอคาซากิซึ่งอาศัยพลังงานจาก ATP ในการเกิดพันธะทางเคมีนี้
โดยสรุปก็คือ การสร้างชิ้นส่วนผสมของไรโบนิวคลีโอไทด์ -5-ไตรฟอสเฟท (ATP GTP CTP และ UTP) ด้วยนั่นคือ การสร้างชิ้นส่วนโอคาซากิต้องการชิ้นส่วนของอาร์เอ็นเอสั้นๆ เป็นไพรเมอร์โดยใช้เอ
นไซม์ไพรเมส และอาศัยดีเอ็นเอพอลีเมอเรสชนิดในการสร้างดีเอ็นเอสายใหม่ต่อจากไพรเมอร์นั่นเอง
สรุปคุณสมบัติของการเป็นสารพันธุกรรมมีดังต่อไปนี้
1.เป็นแหล่งที่เก็บรวบรวมข้อมูลทางพันธุกรรมของสิ่งมีชีวิตซึ่งจะอยู่ในโครงสร้างที่มั่นคงถาวรไม่มีการเปลี่ยนแปลงง่าย ๆ ข้อมูลทางพันธุกรรมเหล่านี้จะเป็นข้อมูลที่เกี่ยวข้องกับโครงสร้าง หน้าที่ การทำงาน ก
ารพัฒนา และการเพิ่มจำนวนของเซลล์สิ่งมีชีวิตนั้น ๆ
2.สามารถเพิ่มจำนวนตัวเอง (self-replication) และถ่ายทอดจากรุ่นหนึ่งไปยังรุ่นต่อ ๆ ไปได้จึงมีผลทำให้เซลล์รุ่นต่อ ๆ ไปนั้นมีข้อมูลทางพันธุกรรมแบบเดียวกันกับเซลล์รุ่นก่อน
3.เกิดการเปลี่ยนแปลงที่เรียกว่า mutation ได้ทำให้เกิดการเปลี่ยนแปลงในข้อมูลทางพันธุกรรมมีผลให้มีการเปลี่ยนแปลงปรับตัวของสิ่งมีชีวิตนั้น เกิดวิวัฒนาการขึ้นได้ เกิดความหลากหลายในสิ่งมีชีวิต
4.ควบคุมการแสดงออกซึ่งพบว่าควบคุมได้ทั้งทางตรงโดยการควบคุมการสังเคราะห์โปรตีนชนิดต่าง ๆ โดยผ่านกระบวนการพิมพ์รหัส (transcription) และแปลรหัส (translation) เรียกว่ากระบวนการสังเครา
ะห์โปรตีน (protein synthesis) ส่วนการควบคุมทางอ้อมโดยการควบคุมการทำงานของยีนอื่น ๆ ได้แก่ระบบ operon ที่พบใน E. coli เป็นต้น ที่เรียกว่า regulation of gene action